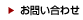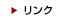研究交流
パイオニアファクターSOX4による肝細胞可塑性の制御機構
勝田 毅
東京大学 大学院工学系研究科 化学システム工学専攻
はじめに
成体肝臓において肝細胞がリプログラミングされるという報告がなされて,10年余りが経った. 2008年に西川祐司先生が解説されているように[1],肝細胞が培養条件下で可塑性を示すことはそれよりも以前から知られていた[2–4].また,2005年には既にこのような現象がin vivoで起こることが報告されている[5].しかし,この報告はDPPIV陽性のラット肝細胞をDPPIV陰性のレシピエント障害肝に移植して作製したキメラ肝ラットを用いた細胞追跡技術に基づくものであった.そのため,人工的な条件でのみ観察される現象ではないかとの懐疑的な見方もあった.その後,lineage tracing研究が進み,キメラ化を伴わない「生理的条件」下でも肝細胞のリプログラミングが改めて確認された[6].その後の10年余りでメカニズム研究が進み,Notch,Hippo/Yap,TGFβ,IL6/STAT3などのシグナル経路が重要であることがわかってきた[6–10].
エピゲノムの観点からの理解は遅れていたが,最近になって徐々に研究が進んできた.2019年のLiらの報告に始まり,私たちも2021年に,クロマチンのリモデリングが肝細胞リプログラミングにおいて重要であることを報告した [11,12].しかし,そのような変化がどのように胆管特異的なものへと方向付けられるのかはわかっていなかった.私たちは最近,転写因子SOX4がリプログラミング初期に重要な役割を果たすことを明らかにしたので[13],本稿ではその研究内容をご紹介したい.
パイオニアファクターに着目したリプログラミングメカニズムの探索
エピジェネティックな変化を先導する因子として,私たちは転写因子の一種であるパイオニアファクターに着目した.一般的な転写因子は,開いたクロマチン領域にあるモチーフにしか結合できない.一方パイオニアファクターは,閉じた領域のモチーフにも結合でき,その領域をこじ開ける能力をもつ[14].完全にクロマチンを開くには,SWI/SNFのようなクロマチンリモデリング因子の助けが必要となるが,そのきっかけづくりでパイオニアファクターが重要な役割を果たす.実際に,パイオニアファクター活性がiPS細胞をはじめとするin vitroのリプログラミングに使われていることもわかってきた.山中4因子のうちOCT4, SOX2, KLF4の3つがパイオニアファクターとして機能していたのである.線維芽細胞では,多能性に関わる遺伝子領域は閉じていて転写活性をもたないが,OCT4, SOX2, KLF4がそれらの領域を開くことで,多能性獲得のプライミング機構として働いていることが明らかになった[15,16].
このような知見を背景に,肝細胞が障害を受けると,何らかのパイオニアファクターの発現が誘導され,本来閉じた領域にあった胆管遺伝子領域が開かれ,リプログラミングの開始が誘導されるとの仮説を立てた.私たちは,先行研究でのATAC-seqとRNA-seqの解析から,リプログラミング後にクロマチンの開き具合(アクセシビリティ)が増加し,かつ胆管表現型と相関をもつ領域に,SOX転写因子のモチーフが高頻度に出現することを見出していた[12](図1A).また,リプログラミングの各段階での遺伝子発現を見たところ,Sox4とSox9の2つだけが,肝障害を受けた細胞で顕著に上昇していたことから,これらの遺伝子のいずれか,または両方がパイオニアファクターとして機能するとの作業仮説に落とし込んだ.
図1. 胆管リプログラミングを誘導するパイオニアファクター候補の探索
(A) リプログラミング過程で発現誘導され,新たにオープンになった遺伝子領域において高頻度で見つかったモチーフ.右下の各種SOXモチーフの具体的な配列を見てもわかるように,コアとなる配列C(T/A)TTGTはいずれにも共通している.そのため,モチーフ解析だけから,具体的にどの因子が結合していたかを推測することは難しく,実際の発現解析から検討する必要があった.私たちはRNA-seqでリプログラミング過程において発現上昇してくるSox遺伝子として,Sox4とSox9を同定し,パイオニアファクター候補とした.(文献12より改変).
(B) リプログラミング細胞で実際に発現が認められたSox4, Sox9遺伝子,およびその両方を発現させた肝細胞を用いて行ったqRT-PCRの結果.EVはempty vectorを示す.(文献13より改変).
SOX4による胆管表現型の発現誘導
仮説を検証するために,アデノ随伴ウイルスを用いてSox4,Sox9,および両方を肝細胞特異的にin vivoで発現させる実験を行った.その結果,Sox4およびSox4/9両方を発現させた群で,Cd24aやProm1をはじめとするリプログラミング初期のマーカー遺伝子の発現誘導が確認できた(図1B).一方,Sox9単独の発現ではリプログラミングの兆候はみられず,またSox4/9の共発現で相乗・相加的な効果はみられなかった(図1B).そのため,これ以降の研究ではSOX4に焦点を絞ることとした.なお,Sox4発現によるEpcamなどのリプログラミング後期のマーカー遺伝子の発現誘導は限定的で,さらに成熟胆管細胞のマーカー遺伝子であるKrt19などの発現誘導に至っては全く見られなかった.このことから,Sox4の影響はリプログラミング初期から中期にかけての範囲に限られると考えられる.
続いてSOX4がパイオニアファクターとしての機能をもつかどうかを検証した.これには,SOX4モチーフを含む合成オリゴDNAをヒストン8量体に巻き付ける形で,in vitroで再構築したヌクレオソームへの結合能を持つかどうかで評価した[17](図2A).DNAがヌクレオソームと複合体を形成すると結合モチーフが埋もれた形となり,閉じたクロマチンとよく似た構造をとる.そのため,パイオニアファクターでない転写因子はそのようなモチーフには結合できなくなる.この原理を利用し,HEK293T細胞で産生したSOX4タンパクを精製し,SOX4結合モチーフを含むヌクレオソームと反応させ,ゲルシフトアッセイ(EMSA)を行ったところ, SOX4がヌクレオソームに結合することが確かめられた(図2B).
図2. ゲルシフトアッセイによるパイオニアファクター活性の評価
(A) ヒストン分子からin vitroで再構築したヌクレオソームを用いたゲルシフトアッセイの概要.ここではパイオニアファクターとして知られるFOXA1がその標的となるアルブミンプロモーターに結合する様子が示されている.(文献17より改変)
(B) SOX結合モチーフを含むLIN28BオリゴDNA断片の模式図(上,文献16より改変).下に,実際のSOX4のパイオニアファクター活性の評価を行った結果を示す.(文献13より改変)
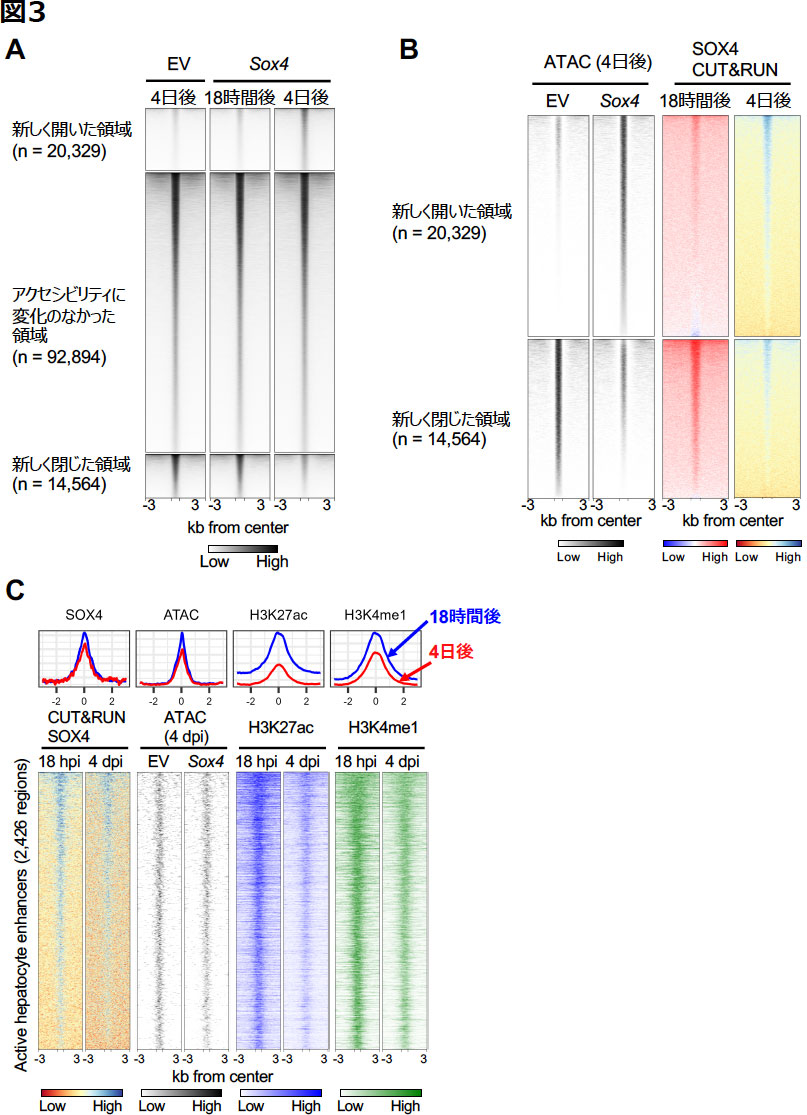
続いて,SOX4の結合が,肝細胞のクロマチン状態にどのような影響を与えるかを検証していった.まずATAC-seqによってSOX4発現前後でのオープンクロマチンの変化を比較した(図3A).SOX4発現後の肝細胞でオープンな領域のうち,約18%がSOX4の発現後特異的なものであることが分かった.このような新たに開いた領域がSOX4の結合によるものかどうかを見るために,CUT&RUN-seqによってSOX4の結合箇所をゲノムワイドに解析した.その結果,胆管の表現型が見られるようになる発現4日後の時点において,新たに開いた領域の多くでSOX4の結合が認められた(図3B上).
図3. 肝細胞におけるSox4遺伝子の発現によるクロマチン変化とSOX4の結合プロファイル
(A) Sox4遺伝子発現後のオープンクロマチンプロファイルの変化を,18時間後および4日後にATAC-seqで評価した.
(B) 新しく開いた領域と新しく閉じた領域でSOX4の結合が見られるかどうか,18時間後と4日後の2時点でCUT&RUN-seqによって評価した.
(C) 肝細胞特異的なエンハンサー領域における,ATAC-seqおよび,活性化ヒストン翻訳後修飾H3K27acとH3K4me1のCUT&RUN-seq結果.18 hpi = 18時間後,4 dpi = 4時間後.
(文献13より改変)
SOX4による肝細胞表現型の抑制
パイオニアファクターが閉じた領域にも結合できることを考慮し,胆管表現型が見られる前の,発現18時間後という極めて早い時期でもSOX4のCUT&RUN-seqを行った.その結果,予想に反し,18時間後でのSOX4の結合は,元の肝細胞では開いていてSOX4発現4日後に閉じることになる領域にリッチであることが判明した(図3B下).このことからSOX4が,胆管の表現型獲得の前に,肝細胞の表現型の喪失を誘導している可能性が見えてきた.
さらに興味深いことに,18時間後のSOX4の結合が,肝細胞特異的なエンハンサー領域で顕著にみられ,またこれらのエンハンサー活性が4日後に低下することを見出した(図3C).プロモーターはその下流遺伝子の発現の有無に関わらず常にオープンな状態にあることが多いのに対し,エンハンサーの開き具合は標的遺伝子の発現とより強い相関を示す[18].このことから,エンハンサーは細胞のアイデンティティを規定する重要な因子と考えられている.ATAC-seqの結果から,発現後18時間でのSOX4の結合が見られた肝細胞エンハンサーでは,4日後にアクセシビリティの顕著な低下がみられることがわかった.さらに,ヒストンの翻訳後修飾をCUT&RUN-seqで見たところ,エンハンサー活性の指標であるH3K27acやH3K4me1のレベルもこれらの領域で低下していた(図3C).
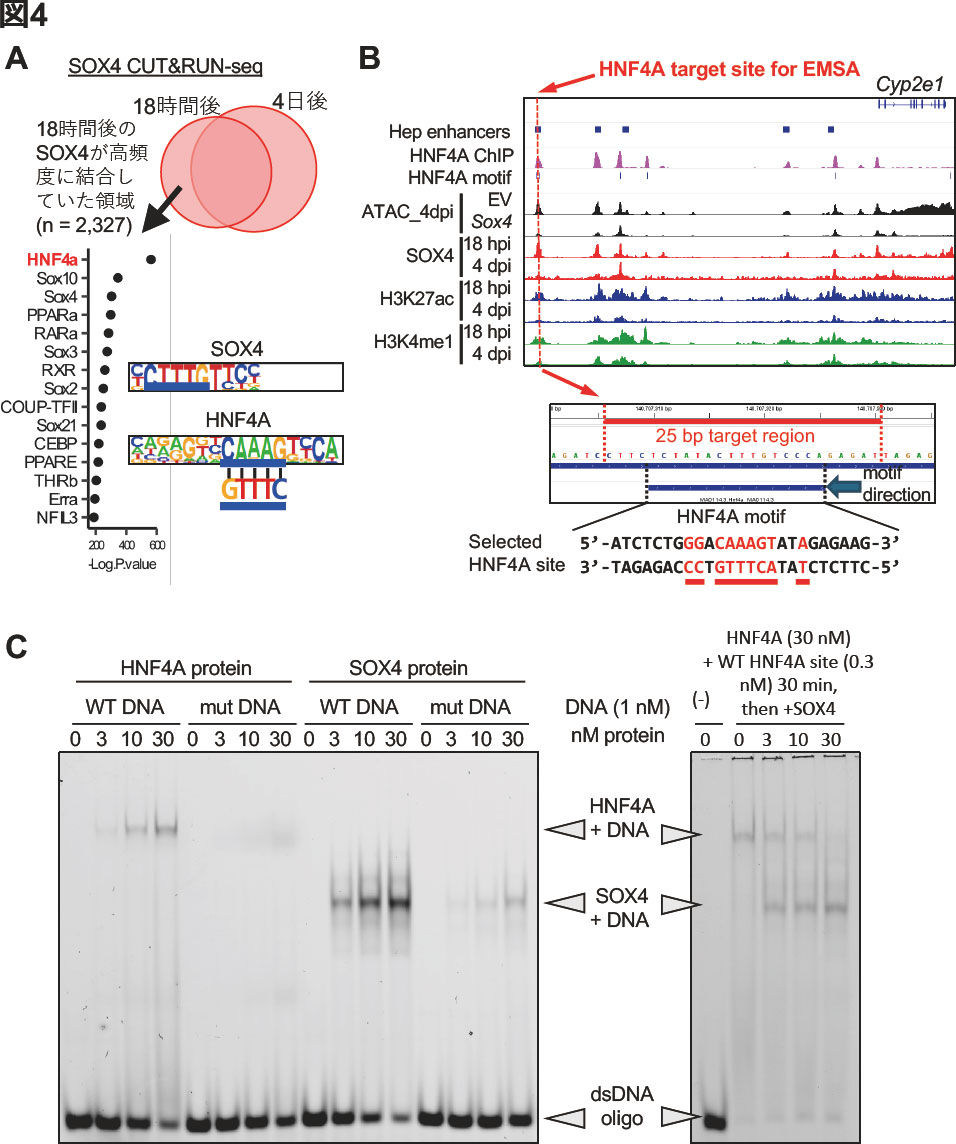
SOX4が肝細胞エンハンサーに結合すると,なぜアクセシビリティが低下するのかをさらに探っていった.まずSOX4のCUT&RUN-seqにより,発現18時間後と4日後で結合領域がどのように変わるのかを検討した.18時間後でより顕著にSOX4の結合が見られる領域についてモチーフ解析を行ったところ,驚いたことに最上位のヒットがSOXモチーフではなく,HNF4Aモチーフであった(図4A).また,モチーフの配列を注意深く見ると,コアとなる配列がSOX4とHNF4Aとで顕著にオーバーラップしていることに気が付いた(図4A).
このことから,SOX4がHNF4Aのモチーフを認識してその領域を乗っ取り,その結果,肝細胞エンハンサーの不活性化が起こったのではないかと考えた.そこで,今度はHNF4Aの標的遺伝子であるCyp2e1のエンハンサーに着目し,HNF4AとSOX4との間で結合モチーフの奪い合いが起こるかどうかを検討することにした.Cyp2e1のエンハンサーは複数あるが,特にHNF4Aモチーフを含む一つに照準を絞った(図4B).そのモチーフを含む25 bpの合成オリゴDNAを用いてゲルシフトアッセイを行ったところ,SOX4が配列特異的にエンハンサーDNAに結合することが確認された(図4C).さらに,DNA断片にあらかじめHNF4Aタンパクを結合させておき,それにSOX4タンパクを様々な濃度で添加するという競合実験を行ったところ,同じ濃度に達すると完全にSOX4の結合に置き換わることが判明した(図4C).
図4. SOX4は,HNF4Aが結合したエンハンサーを標的とし,その結合モチーフを乗っ取る.
(A) SOX4発現直後に,より顕著であった結合領域でモチーフ解析を行った結果.
(B) HNF4Aの標的遺伝子であるCyp2e1のエンハンサーで,HNF4Aのモチーフがあるものを一つ選び,モチーフを含む25 bpのオリゴDNAを合成し,ゲルシフトアッセイ(EMSA)に用いた.
(C) ゲルシフトアッセイの結果.HNF4A,SOX4ともに標的となるHNF4Aモチーフの配列特異的に結合していることがわかる(左).また,HNF4AとオリゴDNAからあらかじめタンパク/DNA複合体を形成させておいて(正常肝細胞の核内の状態を想定),そこにSOX4を添加していき,競合が起こることを確認した(右).
(文献13より改変)
おわりに
パイオニアファクターが細胞のリプログラミングにおいて重要であるとの考えは,in vitroでは既に確立されている.しかし,外的因子との相互作用が重要と考えられるin vivoの環境でもこのシナリオが成り立つのかどうかは不明であった.私たちの研究は,そのような極めて雑多なin vivo環境でもパイオニアファクターが重要な役割を果たすことを示した例である.
また,転写を活性化するだけと考えていたパイオニアファクターに,転写抑制の働きがあるという発見は,私たちにとって予期せぬ成果となった.私たちの報告以前にも,パイオニアファクターによる抑制の報告がいくつかあったが,その数は少なく,ほとんど未開拓の研究領域であった[19,20].ところが最近になって,私たちの例も含め報告数が増えつつある[13,21–23].このトピックにもし興味を持たれた方がいらっしゃれば,私たちが最近寄稿したミニレビュー論文も合わせてお読みいただければ幸いである[24].
謝辞
本研究はペンシルベニア大学のBen Stanger教授とKen Zaret教授の下で行ったものです.この場を借りて御礼申し上げます.また,本稿執筆の機会を与えてくださった汐田剛史先生と田中稔先生に感謝申し上げます.
参考文献
- 西川祐司. (2008). 肝障害と幹細胞 ― 肝修復における幹細胞の役割. 肝細胞研究会 研究交流. http://hepato.umin.jp/kouryu/kouryu04.html
- Nishikawa, Y., Doi, Y., Watanabe, H., Tokairin, T., Omori, Y., Su, M., Yoshioka, T., & Enomoto, K. (2005). Transdifferentiation of mature rat hepatocytes into bile duct-like cells in vitro. American Journal of Pathology, 166(4), 1077–1088. https://doi.org/10.1016/S0002-9440(10)62328-0
- Michalopoulos, G. K., Bowen, W. C., Mulè, K., Lopez-Talavera, J. C., & Mars, W. (2002). Hepatocytes undergo phenotypic transformation to biliary epithelium in organoid cultures. Hepatology (Baltimore, Md.), 36(2), 278–283. https://doi.org/10.1053/jhep.2002.34858
- Nishikawa, Y., Tokusashi, Y., Kadohama, T., Nishimori, H., & Ogawa, K. (1996). Hepatocytic cells form bile duct-like structures within a three-dimensional collagen gel matrix. Experimental Cell Research, 223(2), 357–371. https://doi.org/10.1006/excr.1996.0091
- Michalopoulos, G. K., Barua, L., & Bowen, W. C. (2005). Transdifferentiation of rat hepatocytes into biliary cells after bile duct ligation and toxic biliary injury. Hepatology (Baltimore, Md.), 41(3), 535–544. https://doi.org/10.1002/hep.20600
- Yanger, K., Zong, Y., Maggs, L. R., Shapira, S. N., Maddipati, R., Aiello, N. M., Thung, S. N., Wells, R. G., Greenbaum, L. E., & Stanger, B. Z. (2013). Robust cellular reprogramming occurs spontaneously during liver regeneration. Genes & Development, 27(7), 719–724. https://doi.org/10.1101/gad.207803.112
- Yimlamai, D., Christodoulou, C., Galli, G. G., Yanger, K., Pepe-Mooney, B., Gurung, B., Shrestha, K., Cahan, P., Stanger, B. Z., & Camargo, F. D. (2014). Hippo pathway activity influences liver cell fate. Cell, 157(6), 1324–1338. https://doi.org/10.1016/j.cell.2014.03.060
- Tarlow, B. D. D., Pelz, C., Naugler, W. E. E., Wakefield, L., Wilson, E. M. M., Milton, J., Finegold, M. J., & Grompe, M. (2014). Bipotential Adult Liver Progenitors Are Derived from Chronically Injured Mature Hepatocytes. Cell Stem Cell, 15(5), 605–618. https://doi.org/10.1016/j.stem.2014.09.008
- Schaub, J. R., Huppert, K. A., Kurial, S. N. T., Hsu, B. Y., Cast, A. E., Donnelly, B., Karns, R. A., Chen, F., Rezvani, M., Luu, H. Y., Mattis, A. N., Rougemont, A. L., Rosenthal, P., Huppert, S. S., & Willenbring, H. (2018). De novo formation of the biliary system by TGFβ-mediated hepatocyte transdifferentiation. Nature, 557(7704), 247–251. https://doi.org/10.1038/s41586-018-0075-5
- Li, L., Cui, L., Lin, P., Liu, Z., Bao, S., Ma, X., Nan, H., Zhu, W., Cen, J., Mao, Y., Ma, X., Jiang, L., Nie, Y., Ginhoux, F., Li, Y., Li, H., & Hui, L. (2023). Kupffer-cell-derived IL-6 is repurposed for hepatocyte dedifferentiation via activating progenitor genes from injury-specific enhancers. Cell Stem Cell, 30(3), 283-299.e9. https://doi.org/10.1016/j.stem.2023.01.009
- Li, W., Yang, L., He, Q., Hu, C., Zhu, L., Ma, X., Ma, X., Bao, S., Li, L., Chen, Y., Deng, X., Zhang, X., Cen, J., Zhang, L., Wang, Z., Xie, W. F., Li, H., Li, Y., & Hui, L. (2019). A Homeostatic Arid1a-Dependent Permissive Chromatin State Licenses Hepatocyte Responsiveness to Liver-Injury-Associated YAP Signaling. Cell Stem Cell, 25(1), 54-68.e5. https://doi.org/10.1016/j.stem.2019.06.008
- Merrell, A. J., Peng, T., Li, J., Sun, K., Li, B., Katsuda, T., Grompe, M., Tan, K., & Stanger, B. Z. (2021). Dynamic transcriptional and epigenetic changes drive cellular plasticity in the liver. Hepatology (Baltimore, Md.). https://doi.org/10.1002/hep.31704
- Katsuda, T., Sussman, J. H., Ito, K., Katznelson, A., Yuan, S., Takenaka, N., Li, J., Merrell, A. J., Cure, H., Li, Q., Rasool, R. U., Asangani, I. A., Zaret, K. S., & Stanger, B. Z. (2024). Cellular reprogramming in vivo initiated by SOX4 pioneer factor activity. Nature Communications, 15(1), 1–20. https://doi.org/10.1038/s41467-024-45939-z
- Zaret, K. S. (2020). Pioneer Transcription Factors Initiating Gene Network Changes. Annual Review of Genetics, 54, 367–385. https://doi.org/10.1146/annurev-genet-030220-015007
- Soufi, A., Donahue, G., & Zaret, K. S. (2012). Facilitators and impediments of the pluripotency reprogramming factors’ initial engagement with the genome. Cell, 151(5), 994–1004. https://doi.org/10.1016/j.cell.2012.09.045
- Soufi, A., Garcia, M. F., Jaroszewicz, A., Osman, N., Pellegrini, M., & Zaret, K. S. (2015). Pioneer Transcription Factors Target Partial DNA Motifs on Nucleosomes to Initiate Reprogramming. Cell, 161(3), 555–568. https://doi.org/10.1016/j.cell.2015.03.017
- Takizawa, Y., Tanaka, H., Machida, S., Koyama, M., Maehara, K., Ohkawa, Y., Wade, P. A., Wolf, M., & Kurumizaka, H. (2018). Cryo-EM structure of the nucleosome containing the ALB1 enhancer DNA sequence. Open Biology, 8(3). https://doi.org/10.1098/rsob.170255
- Iwafuchi-Doi, M., Donahue, G., Kakumanu, A., Watts, J. A., Mahony, S., Pugh, B. F., Lee, D., Kaestner, K. H., & Zaret, K. S. (2016). The Pioneer Transcription Factor FoxA Maintains an Accessible Nucleosome Configuration at Enhancers for Tissue-Specific Gene Activation. Molecular Cell, 62(1), 79–91. https://doi.org/10.1016/j.molcel.2016.03.001
- Chronis, C., Fiziev, P., Papp, B., Butz, S., Bonora, G., Sabri, S., Ernst, J., & Plath, K. (2017). Cooperative Binding of Transcription Factors Orchestrates Reprogramming. Cell, 168(3), 442-459.e20. https://doi.org/10.1016/j.cell.2016.12.016
- Sekiya, T., & Zaret, K. S. (2007). Repression by Groucho/TLE/Grg Proteins: Genomic Site Recruitment Generates Compacted Chromatin In Vitro and Impairs Activator Binding In Vivo. Molecular Cell, 28(2), 291–303. https://doi.org/10.1016/j.molcel.2007.10.002
- Thompson, J. J., Lee, D. J., Mitra, A., Frail, S., Dale, R. K., & Rocha, P. P. (2022). Extensive co-binding and rapid redistribution of NANOG and GATA6 during emergence of divergent lineages. Nature Communications, 13(1), 1–18. https://doi.org/10.1038/s41467-022-31938-5
- Yang, Y., Gomez, N., Infarinato, N., Adam, R. C., Sribour, M., Baek, I., Laurin, M., & Fuchs, E. (2023). The pioneer factor SOX9 competes for epigenetic factors to switch stem cell fates. Nature Cell Biology, 25(8), 1185–1195. https://doi.org/10.1038/s41556-023-01184-y
- Matsui, S., Granitto, M., Buckley, M., Ludwig, K., Koigi, S., Shiley, J., Zacharias, W. J., Mayhew, C. N., Lim, H. W., & Iwafuchi, M. (2024). Pioneer and PRDM transcription factors coordinate bivalent epigenetic states to safeguard cell fate. Molecular Cell, 84(3), 476-489.e10. https://doi.org/10.1016/j.molcel.2023.12.007
- Katsuda, T., Sussman, J. H., Zaret, K. S., & Stanger, B. Z. (2024). The yin and yang of pioneer transcription factors: Dual roles in repression and activation. BioEssays, July, 1–6. https://doi.org/10.1002/bies.202400138